Obiettivo n.1
Obiettivo n.1 del progetto di ricerca con la Fondazione Matarelli è lo sviluppo di database.
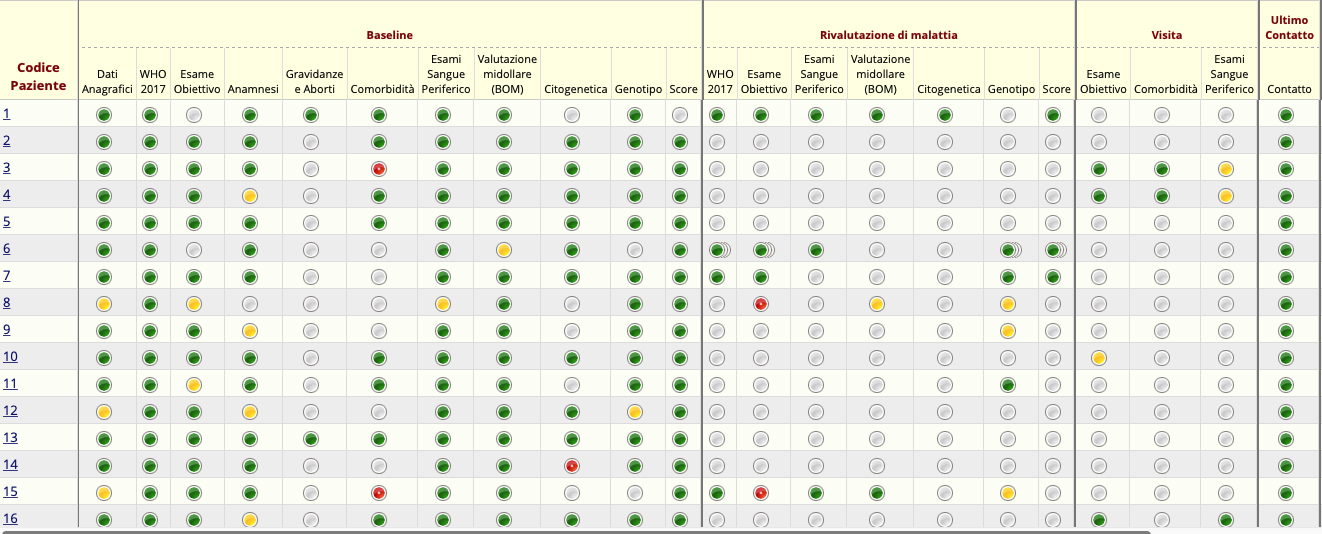
Nell’anno 2018 è stato portato a termine il progetto di un database web-based (RedCap) per lo studio delle neoplasie mieloidi croniche in Ematologia a Varese. Ad oggi il database include 809 pazienti con mielofibrosi, trombocitemia e policitemia. La Figura 1 riporta la configurazione (parziale) del database.
Figura 1: database REDCap
Il progetto sarà condiviso con l’Ematologia dell’Istituto Humanitas con lo scopo di incrementare la casistica e generare studi collaborativi. In questa collaborazione saranno inclusi anche pazienti con leucemia mieloide acuta e sindromi mielodisplastiche.
Inoltre, il database internazionale sulla mielofibrosi post-policitemia vera e posttrombocitemia essenziale, sviluppato nel 2016, è stato implementato con dati genomici di Next Generation Sequencing.
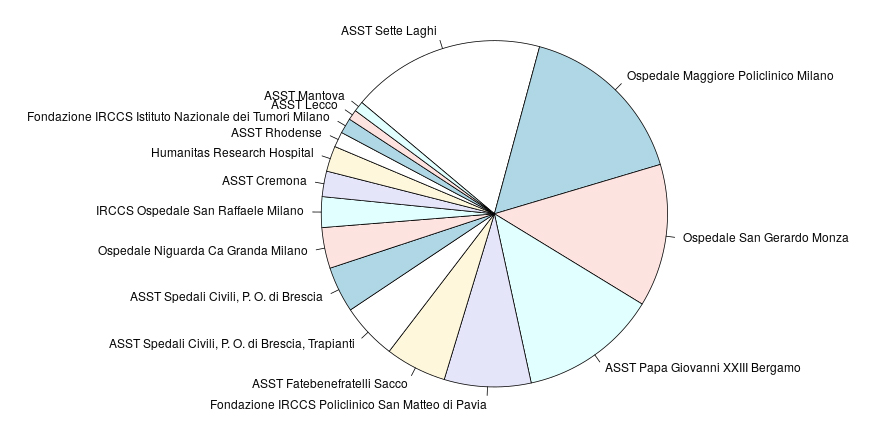
Un ulteriore database (Figura 2) è stato realizzato per lo studio dell’impego di ruxolitinib nella mielofibrosi (RUXO-REL trial). Lo studio prospettico ha incluso 17 centri della Rete Ematologica Lombarda (REL) (Figura 3). Questo è stato la base per uno studio in corso su ruxolitinib come Real World Data per generare Real World Evidence.
Figura 2: database RUXO-REL
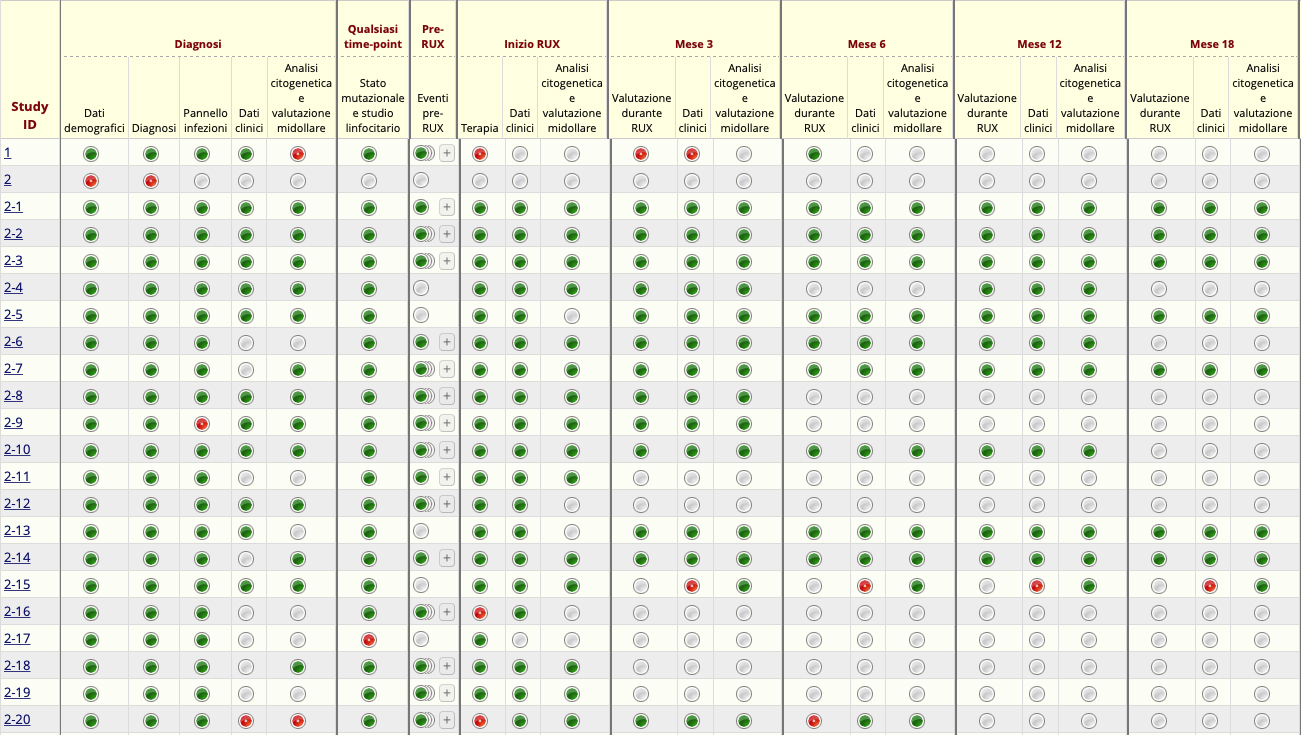
Figura 3: Centri Partecipanti allo studio RUXO-REL
Obiettivo n.2
Obiettivo n.2 del programma di ricerca è quello di valutare la presenza di mutazioni driver e mutazioni addizionali in pazienti affetti da neoplasie mieloidi con tecnologia NGS (Next-Generation Sequencing). Per tale finalità stiamo utilizzando strumentazione MiniSeqTM System, disponibile in Ateneo per la ricerca ematologica.
Questa tecnologia, che permette il sequenziamento di molti geni contemporaneamente e a costi inferiori rispetto all’approccio tradizionale
(sequenziamento di Sanger), è stata scelta per identificare le varianti somatiche dei geni coinvolti nelle neoplasie mieloidi.
Lo studio dell’assetto mutazionale di questi pazienti è necessario per ipotizzare i meccanismi molecolari alla base della patologia, per stratificare i pazienti in classi di rischio e per identificare una correlazione genotipo-fenotipo clinico. Nel 2018 abbiamo finalizzato la piattaforma NGS per le neoplasie mieloidi inizialmente con il sistema Illumina e a seguire con Sophia Genetics. Sono state inoltre acquisite alcune strumentazioni e prodotti accessori necessari per effettuare la preparazione dei campioni e per il sequenziamento. Abbiamo raccolto materiale genetico sui pazienti previo consenso informato. Il sangue periferico dei pazienti con neoplasie mieloidi, tramite gradiente di densità con Ficoll, è stato separato lo stesso giorno del prelievo nelle diverse popolazioni cellulari; dalle cellule polimorfonucleate è stato estratto il DNA rappresentativo della patologia mentre dalla frazione di cellule mononucleate sono stati selezionati positivamente i linfociti T mediante biglie magnetiche coniugate con anticorpi anti-CD3 (Dynabeads CD3, Invitrogen). Il DNA estratto dai linfociti T è stato utilizzato come controllo costituzionale. Il confronto tra lo stato mutazionale del DNA somatico e di quello costituzionale permette l’identificazione delle varianti somatiche rilevanti per la malattia.
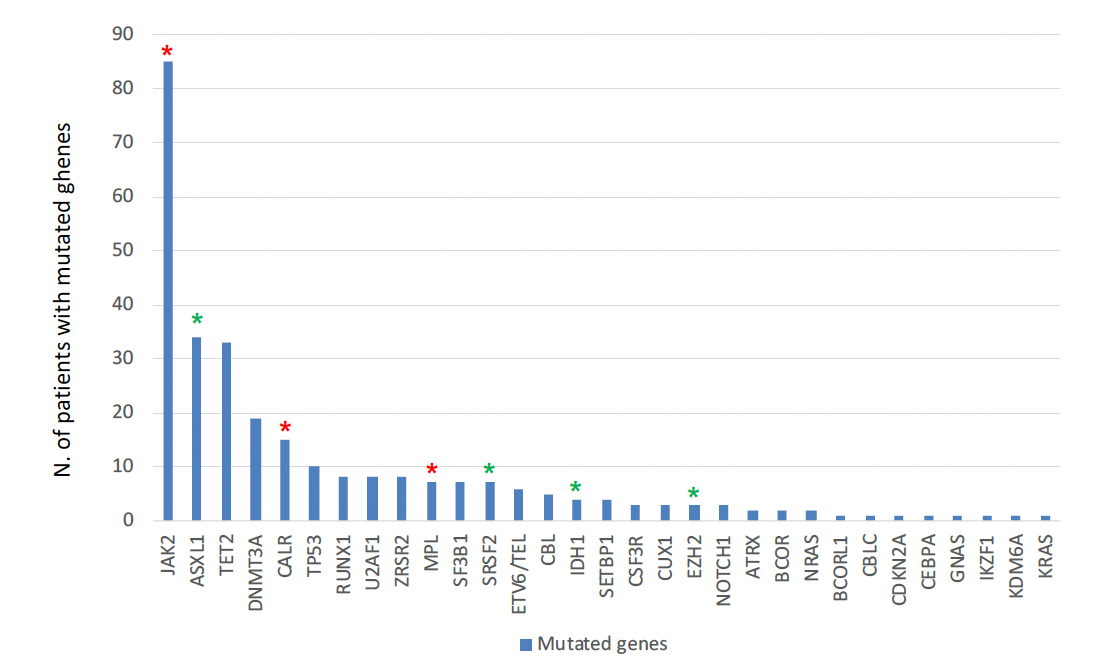
La Figura 4 mostra le varianti alleliche identificate in una prima coorte di 152 pazienti con neoplasie mieloidi.
Figura 4: mutazioni in NGS in pazienti con neoplasie mieloidi
Obiettivo N.3
Obiettivo finale del progetto è pubblicare informazioni nuove. Nel 2018 il contributo scientifico si è svolto attraverso la pubblicazione di diversi
lavori sull’argomento.
- Direct-Acting Antivirals in Hepatitis C Virus-Associated Diffuse Large B-cell Lymphomas.
Merli M, Frigeni M, Alric L, Visco C, Besson C, Mannelli L, Di Rocco A, Ferrari A, Farina L, Pirisi M, Piazza F, Loustaud-Ratti V, Arcari A, Marino D, Sica A, Goldaniga M, Rusconi C, Gentile M, Cencini E, Benanti F, Rumi MG, Ferretti VV, Grossi P, Gotti M, Sciarra R, Tisi MC, Cano I, Zuccaro V, Passamonti F, Arcaini L.
Oncologist. 2018 Dec 14. pii: theoncologist.2018-0331. - Gender effect on phenotype and genotype in patients with postpolycythemia vera and post-essential thrombocythemia myelofibrosis: results from the MYSEC project.
Barraco D, Mora B, Guglielmelli P, Rumi E, Maffioli M, Rambaldi A, Caramella M, Komrokji R, Gotlib J, Kiladjian JJ, Cervantes F, Devos T, Palandri F, De Stefano V, Ruggeri M, Silver RT, Benevolo G, Albano F, Merli M, Pietra D, Barbui T, Rotunno G, Cazzola M, Giorgino T, Vannucchi AM, Passamonti F.
Blood Cancer J. 2018 Sep 21;8(10):89. - Chronic myeloproliferative neoplasms in the elderly.
Maffioli M, Orlandi E, Passamonti F.
Eur J Intern Med. 2018 Dec;58:33-42. - Phenotype variability of patients with post polycythemia vera and post essential thrombocythemia myelofibrosis is associated with the time to progression from polycythemia vera and essential thrombocythemia.
Mora B, Giorgino T, Guglielmelli P, Rumi E, Maffioli M, Rambaldi A, Caramella M, Komrokji R, Gotlib J, Kiladjian JJ, Cervantes F, Devos T, Palandri F, De Stefano V, Ruggeri M, Silver RT, Benevolo G, Albano F, Cavalloni C, Barraco D, Pietra D, Barbui T, Rotunno G, Vannucchi AM, Passamonti F.
Leuk Res. 2018 Jun;69:100-102. - The role of JAK2 inhibitors in MPNs 7 years after approval.
Passamonti F, Maffioli M.
Blood. 2018 May 31;131(22):2426-2435. - Value of cytogenetic abnormalities in post-polycythemia vera and postessential thrombocythemia myelofibrosis: a study of the MYSEC project.
Mora B, Giorgino T, Guglielmelli P, Rumi E, Maffioli M, Rambaldi A, Caramella M, Komrokji R, Gotlib J, Kiladjian JJ, Cervantes F, Devos T, Palandri
F, De Stefano V, Ruggeri M, Silver RT, Benevolo G, Albano F, Cavalloni C, Barraco D, Merli M, Pietra D, Casalone R, Barbui T, Rotunno G, Cazzola M,
Vannucchi AM, Passamonti F.
Haematologica. 2018 Sep;103(9):e392-e394. - Blast phase myeloproliferative neoplasm: Mayo-AGIMM study of 410 patients from two separate cohorts.
Tefferi A, Mudireddy M, Mannelli F, Begna KH, Patnaik MM, Hanson CA, Ketterling RP, Gangat N, Yogarajah M, De Stefano V, Passamonti F, Rosti V, Finazzi MC, Rambaldi A, Bosi A, Guglielmelli P, Pardanani A, Vannucchi AM.
Leukemia. 2018 May;32(5):1200-1210.



